In der Medizin fallen viele Daten an, etwa bei der Computertomographie. Auf dem Weg zur personalisierten Medizin sind sie wichtig. Verfahren der Künstlichen Intelligenz, wie etwa das maschinelle Lernen, lernen mit ihnen dazu und helfen, Diagnosen und Therapien künftig individuell zu zuschneiden. Solche Systeme sind derzeit aber noch mit Unsicherheiten behaftet. Ein Forscherteam aus Kaiserslautern und Leipzig arbeitet an einem System, das medizinische Daten automatisch analysiert und visualisiert, auch deren Unsicherheiten. Auf der Medizintechnikmesse Medica in Düsseldorf stellt es seine Technik vom 14. bis 17. November am Forschungsstand Rheinland-Pfalz (Stand E80, Halle 3) vor.
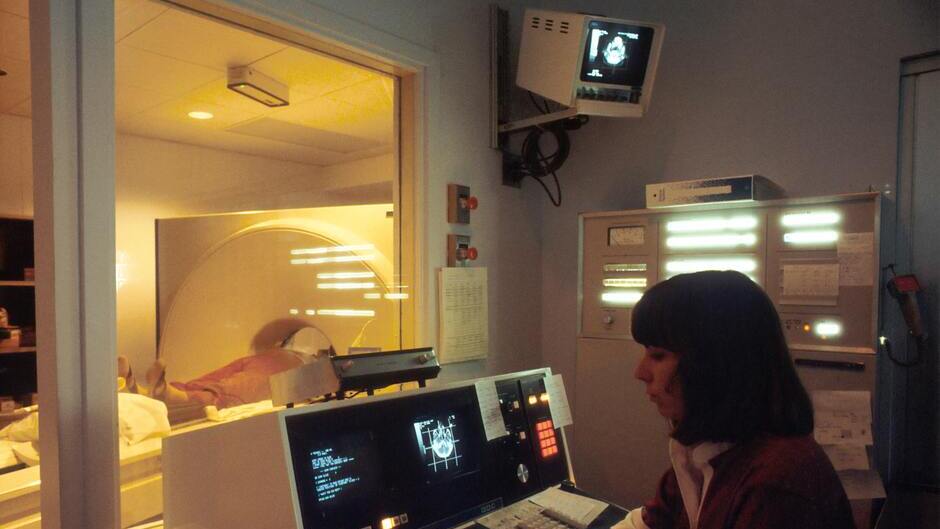
Beim Schlaganfall ist Eile geboten. Mithilfe von Aufnahmen aus dem Computertomographen (CT) können Ärzte schnell feststellen, an welcher Stelle im Gehirn es zu einem Blutgerinnsel gekommen ist und welche Behandlung sinnvoll ist. Solche bildgebenden Verfahren spielen in der Medizin eine wichtige Rolle. Sie kommen auch bei anderen Bereichen, etwa vor Operationen, zum Einsatz. So helfen Bilder von Magnetresonanztomografien (MRT) Chirurgen beispielsweise vor einer Operation, einen Eingriff zu planen.
All diesen Techniken gemein ist, dass jede Menge Daten anfallen. „Sie automatisch zu analysieren und zu visualisieren, ist ein wichtiger Schritt auf dem Weg zur personalisierten Medizin“, sagt Dr. Christina Gillmann, Informatikerin an der Universität Leipzig. „Dieser Bereich hat in den letzten Jahren stark an Bedeutung gewonnen.“ Möglich machen das KI-Verfahren wie das maschinelle Lernen und neuronale Netzwerke. Sie lernen anhand von Daten dazu, mit denen sie trainiert beziehungsweise „gefüttert“ werden. Zum Beispiel aus CT-Bilddaten, die ein Arzt zuvor bearbeitet hat. Auf diese Weise fließen technische Informationen, aber auch medizinische Erfahrung ein. Dabei gilt: Je mehr Daten diese Verfahren auswerten können, desto besser werden die Ergebnisse.
Solche Technologien könnten in ein paar Jahren im Klinikalltag zum Einsatz kommen, um etwa personalisierte Diagnosen und Therapien zu ermöglichen. Allerdings stecken sie noch in den Kinderschuhen. „Jeder medizinische Fall muss einzeln trainiert werden. Die Daten müssen vorab einzeln aufbereitet werden, was sehr aufwändig ist“, nennt Robin Maack aus der Arbeitsgruppe „Computer Graphics and Human Computer Interaction“ an der Technischen Universität Kaiserslautern als Problem. Dazu müssen Ärzte beispielsweise bei jedem medizinischen Fall die Daten einzeln „labeln“. „Das bedeutet beispielsweise, wenn ein Netzwerk trainieren soll, einen Tumor automatisch zu erkennen, müssen bei hunderten Bildern mit bekannten Tumoren diese per Hand eingezeichnet werden, damit das neurale Netzwerk eine Grundlage hat, mit der es lernen kann“, erklärt Gillmann.
Maack fährt fort: „Hinzu kommt, dass es keine einheitlichen Schnittstellen gibt, mit denen trainierte Netzwerke behandelt, geladen und genutzt werden können. Aber auch bei Unsicherheiten in den Datenlagen; sei es bei Trainingsdatensätzen oder bei genutzten Modellen; dafür gibt es keine standardisierten Vorgaben, wie Mediziner damit umgehen sollen.“
Solche Unsicherheiten treten zum Beispiel bei Läsionen auf. Dabei handelt es sich um bestimmte Bereiche im Gehirn, die bei einem Schlaganfall durch den Verschluss von Gefäßen im Gehirn nicht mehr ausreichend oder gar nicht mit Sauerstoff versorgt werden. Sie sind nicht mehr leistungsfähig. Der Kern der Läsion ist oft gut zu erkennen, am Rand gibt es jedoch meist keine klare Abgrenzung und Regionen, bei denen sich selbst Ärzte nicht einig sind, ob sie nun als Läsion zu klassifizieren sind oder nicht. Letztendlich braucht es hier die medizinische Erfahrung, wie damit umzugehen ist.
Hier setzt die Arbeit von Gillmann und Maack an. Das Team um die beiden entwickelt derzeit ein einheitliches System, um medizinische Bilddaten zu verarbeiten, auszuwerten und deren Unsicherheiten zu visualisieren. Es trägt den Namen GUARDIAN, zu Deutsch „Hüter“. Ihre Technik gestalten die Forscher so, dass sie einfach in der Handhabung ist. „Hier können Kliniken zum Beispiel ihre trainierten neuralen Netze laden und diese mit zur Verfügung gestellten aufbereiteten Daten kombinieren, etwa bei Aufnahmen zu einem Schlaganfall.“
Das System wertet die Daten aus und visualisiert die Ergebnisse. „Das geschieht automatisch, ohne dass IT-Kenntnisse notwendig sind“, so Maack weiter. „Zudem zeigt unsere Technik die Unsicherheiten an.“ Das heißt, die Ärzte können sich diese noch einmal anschauen und bei Bedarf gemeinsam eine Entscheidung treffen, was zum Beispiel im Einzelfall die beste Behandlung ist.
Das System steht als Open-Source-Anwendung frei zur Verfügung. Auf der Messe stellen die beiden Informatiker es vor.
Die Arbeitsgruppe „Computer Graphics and Human Computer Interaction“ von Professor Dr. Hans Hagen an der TU Kaiserslautern forscht schon lange daran, Daten aus Bildgebungsverfahren für die Medizin derart aufzubereiten, dass sie im klinischen Alltag einfach und zuverlässig nutzbar sind.
Der Auftritt der Forscherinnen und Forscher der TU Kaiserslautern bei der Medica wird von Klaus Dosch vom Referat für Technologie und Innovation organisiert. Er ist Ansprechpartner für Unternehmen und vermittelt unter anderem Kontakte zur Wissenschaft.
Kontakt: Klaus Dosch, E-Mail: dosch@rti.uni-kl.de, Tel.: 0631 205-3001
Quelle: TU Kaiserslautern
Symbolbild: National Cancer Institute